Laboratório Estratégico
Utiliza técnicas avançadas de biologia molecular para elucidação de eventos em saúde pública e melhoria da capacidade analítica institucional
O Laboratório Estratégico (LEIAL) foi criado em 2017, e sua função primordial é dar respostas rápidas a eventuais ameaças em saúde pública, uma vez que tem por objetivo desenvolver, implantar e implementar métodos laboratoriais de alta complexidade, para agilização da resposta diagnóstica de doenças infecciosas a nível molecular.
O LEIAL, até o momento, foi capaz de responder às demandas feitas ao Instituto Adolfo Lutz que envolveram técnicas de biologia molecular avançadas, e trabalhou com informações provenientes das diversas instituições que compõem a Coordenadoria de Controle de Doenças, como o Centro de Vigilância Epidemiológica, o Centro de Vigilância Sanitária, o Centro de Referência e Treinamento DST/AIDS e o Instituto Pasteur, colaborando com o monitoramento de agravos emergentes em saúde pública. O exemplo mais recente foi o enfrentamento da emergência da MPOX, cujos primeiros resultados na detecção do vírus, assim como a padronização e o depósito do primeiro sequenciamento foram realizados pelo laboratório.
O avanço das tecnologias de análise e processamento de DNA e RNA com o uso dos sequenciadores de nova geração permitiu que o sequenciamento do genoma completo (WGS) de agentes infecciosos se tornasse acessível e com custos cada vez menores. O WGS fornece novas oportunidades para investigar a evolução de patógenos, mesmo em curtos períodos de tempo, pois permite resolução sem precedentes na discriminação de variantes altamente relacionadas, levando a caracterização de diferenças e similaridades entre isolados a um patamar nunca antes obtido. Em comparação com outros métodos de tipagem genética, o WGS oferece resolução definitiva para investigações na vigilância de surtos e epidemias, atribuição de fontes, estudos genômicos e informações genéticas para a previsão de fenótipos (sorotipagem, resistência antimicrobiana, formação de biofilme, transmissibilidade, patogenicidade e virulência).

A Vigilância Genômica dos vírus causadores da gripe, da COVID-19, da dengue e da febre Chikungunya, assim como de agentes bacterianos como N. meningitidis, Salmonella Typhi e Vibrio cholerae, tem sido utilizada pelo Laboratório Estratégico para determinar a origem dos patógenos e suas relações e o esclarecimento dos surtos e epidemias, principalmente na tentativa de estabelecer uma correlação entre a virulência e o impacto desses agentes na população. Como exemplo, a vigilância genômica de S. Typhi e V. cholerae é realizada atualmente em tempo real, com a realização do sequenciamento dos genomas completos, para identificar, precocemente, a introdução de novos clones em nossa região. No gráfico abaixo são apresentados os agentes que tiveram genoma completo sequenciado pelo laboratório estratégico, excluindo-se o SARS-CoV-2, com mais de 4.000 sequências obtidas.
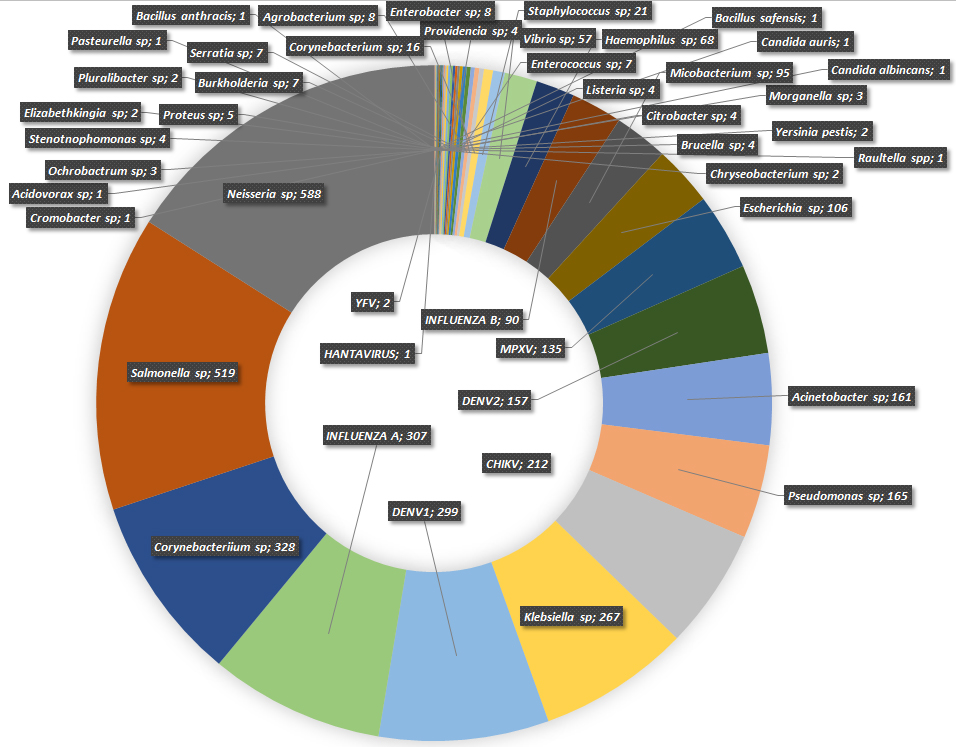
Apesar de muitos avanços, ainda é necessário caminhar para obtenção de maior celeridade e eficácia na produção das informações, que podem permitir o aprimoramento do diagnóstico laboratorial de doenças de interesse epidemiológico, sanitário e ambiental.
Responsável: Claudio Tavares Sacchi
Telefone: (11) 3068-2985
E-mail: labestrategico@ial.sp.gov.br
Produção Científica
Camargo CH, Yamada AY, Souza ARd, Sacchi, CT et al. Genomic characterization of New Delhi metallo-beta-lactamase–producing species of Morganellaceae, Yersiniaceae, and Enterobacteriaceae (other than Klebsiella) from Brazil over 2013–2022. Microbiol Immunol. 2024; 68: 1–5. https://doi.org/10.1111/1348-0421.13100
Ramos JN; Araújo MRB; Sant’Anna LO; Bokermann S; Camargo CH; Prates FD; Sacchi CT; et al. Molecular characterization and whole-genome sequencing of Corynebacterium diphtheriae causing skin lesion. Eur J Clin Microbiol Infect Dis (2023). https://doi.org/10.1007/s10096-023-04706-6
Gräf T; Ferreira, CDN; de Lima GR; de Lima RE; Machado LC; Campos TL; Schemberger MO; Faoro H; Paiva MHS; Bezerra MF; Nascimento V; Souza V; Nascimento F; Mejía M; Silva D; de Oliveira YS; Gonçalves L; Ramos TCA; de Castro DB; Arcanjo AR; Dantas HAP; Presibella MM; Fernandes SB; Gregianini TS; Paz E Silva KM; Sacchi CT et al. Multiple introductions and country-wide spread of DENV-2 genotype II (Cosmopolitan) in Brazil. Virus Evol (2023). https://doi.org/10.1093/ve/vead059
Camargo CH, Yamada AY, de Souza AR, Cunha MPV, Ferraro PSP, Sacchi, CT, dos Santos MB, Campos KR, Tiba-Casas MR, Freire MP, Barretti P. Genomic analysis and antimicrobial activity of β-lactam/β-lactamase inhibitors and other agents against KPC-producing Klebsiella pneumoniae clinical isolates from Brazilian hospitals. Sci Rep 13, 14603 (2023). https://doi.org/10.1038/s41598-023-41903-x
de Souza LM, de Oliveira ID, Sales FCS, da Costa AC, Campos KR, Abbud A, Guerra JM, Borges CSC, Takahashi CPFJ, de Araujo, LJT. Technical comparison of MinIon and Illumina technologies for genotyping Chikungunya virus in clinical samples. J Genet Eng Biotechnol 21, 88 (2023). https://doi.org/10.1186/s43141-023-00536-3
Araújo, MRB, Ramos, JN, de Oliveira Sant’Anna, L, Bokermann S, Santos MBN, Mattos-Guaraldi AL, Azevedo V, Prates FD, Rodrigues DLN, F Aburjaile FF, Sacchi CT, Campos KR et al. Phenotypic and molecular characterization and complete genome sequence of a Corynebacterium diphtheriae strain isolated from cutaneous infection in an immunized individual. Braz J Microbiol (2023). https://doi.org/10.1007/s42770-023-01086-z
Camargo CH, Yamada AY, Souza ARd, Lima MdJdC, Cunha MPV, Ferraro PSP, Sacchi CT, Santos MBNd, Campos KR, Tiba-Casas MR, et al. Genomics and Antimicrobial Susceptibility of Clinical Pseudomonas aeruginosa Isolates from Hospitals in Brazil. Pathogens. 2023; 12(7):918. https://doi.org/10.3390/pathogens12070918
Andrade, TS; Camargo, CH; Campos, KR; Reis, AD; Santos, MBN; Zanelatto, VN; Takagi, EH; Sacchi, CT. Whole genome sequencing of Bacillus anthracis isolated from animal in the 1960s, Brazil, belonging to the South America subclade. Comparative Immunology, Microbiology and Infectious Diseases (2023). https://doi.org/10.1016/j.cimid.2023.102027
Cassiolato, AP; Camargo, CH; Piccoli Cecconi, MC; Christakis, S; Gonçalves, CR; Rodrigues Campos, K; Takenori Higa, F; Andrade Pereira, G; De Moraes, C; Silva de Lemos, AP. Genomic characterization of invasive meningococcal X isolates from Brazil, 1992–2022. Int Microbiol (2023). https://doi.org/10.1007/s10123-023-00325-5
Caterino-de-Araujo, A., Campos, KR, Cabral-de-Oliveira, EC, Rodrigues, AKS, Silva, RX, Azevedo, BV, & Marcusso, RMN. CCR5-¿32, CCR2-64I, SDF1-3’A, and IFNλ4 rs12979860 and rs8099917 gene polymorphisms in individuals with HIV-1, HIV/HTLV-1, and HIV/HTLV-2 in São Paulo, Brazil. Microbes, Infection and Chemotherapy, 3, e1855, 2023 https://doi.org/10.54034/mic.e1855
Kindermann, E. H. S. ; campos, KR ; Caterino-de-Araujo, A . Performance of the tetra-primer PCR technique compared to PCR-RFLP in the search for rs12979860 (C/T) and rs8099917 (T/G) single nucleotide polymorphisms (SNPs) in the IFNL4 gene. REVISTA - INSTITUTO ADOLFO LUTZ, v. 82, p. 1-12, 2023. https://doi.org/10.53393/rial.2023.v82.39195
De Paula, R. A. C. ; Campos, K. R. ; Sacchi, C. T. ; amarante, A. F. ; Taniwaki, N. N. ; Nishina, G. M. N. ; De Oliveira, S. M. P. ; D´Agostini, T. L. ; Prado, W. D. A. ; Froes, M. H. ; Veiga, D. P. B. ; Katz, G. ; Santana, L. M. R. ; Fargetti, S. ; Figueiredo, E. ; Bugno, A. ; ABBUD, A. Informe epidemiológico: primeiros casos confirmados de monkeypox. BEPA. Boletim Epidemiológico Paulista, v. 19, p. 1, 2022. https://periodicos.saude.sp.gov.br/BEPA182/article/view/37805/35777
Maeda, AY; Nogueira, JS; Campos, KR; Camargo, CH; Da-Silva Vasami, F G; Arvigo, APB; Santos, MBN; Abbud, A; Sacchi, CT. Circulation of Chikungunya virus East-Central-South African genotype during the 2020-21 outbreak in São Paulo State, Brazil. Journal of Clinical Virology Plus, v. 2, p. 100070, 2022. https://doi.org/10.1016/j.jcvp.2022.100070
Lemos APS, Gorla MCO, de Moraes C, Willemann MC, Sacchi CT, Fukasawa LO, Camargo CH, Barreto G, Rodrigues DS, Gonçalves MG, Higa FT, Salgado MM, de Moraes JC. Emergence of Neisseria meningitidis W South American sublineage strain variant in Brazil: disease and carriage. J Med Microbiol. 2022 Feb;71(2). doi: https://doi.org/10.1099/jmm.0.001484
Campos KR, Sacchi CT, Abbud A, Caterino-de-Araujo A. SARS-CoV-2 variants in severely symptomatic and deceased persons who had been vaccinated against COVID-19 in São Paulo, Brazil. Rev Panam Salud Publica. 2021 Oct 25;45:e126. doi: https://doi.org/10.26633/RPSP.2021.126.
Marín JEO, Villatoro E, Luna MJ, Barrientos AM, Mendoza E, Lemos APS, Camargo CH, Sacchi CT, Cunha MPV, Galas M, Gabastou JM. Emergence of MDR invasive Neisseria meningitidis in El Salvador, 2017-19. J Antimicrob Chemother. 2021 Apr 13;76(5):1155-1159. doi: https://doi.org/10.1093/jac/dkab010.
Camargo CH, Gonçalves CR, Pagnoca EVRG, Campos KR, Montanha JOM, Flores MNP, Soares MMCN, Binhardi FMT, Ferreira PM, Yu ALF, Carvalhanas TRMP, Abbud A, Bugno A, Sacchi CT. SARS-CoV-2 reinfection in a healthcare professional in inner Sao Paulo during the first wave of COVID-19 in Brazil. Diagn Microbiol Infect Dis. 2021 Dec;101(4):115516. doi: https://doi.org/10.1016/j.diagmicrobio.2021.115516.
Camargo, CH; Goncalves, CR; Pagnoca, EVRG; Campos, KR; Abbud, A; Bugno, A; Caterino-de-Araujo, A; Sacchi, CT. Um ano de pandemia da COVID-19: diversidade genética do SARS-CoV-2 no Brasil. BEPA. Boletim Epidemiológico Paulista (online), v. 18, p. 32-45, 2021. Disponível em: https://periodicos.saude.sp.gov.br/index.php/BEPA182/article/view/35843.
Fukasawa LO, Sacchi CT, Gonçalves MG, Lemos APS, Almeida SCG, Caterino-de-Araujo A. Comparative performances of seven quantitative Reverse-Transcription Polymerase Chain Reaction assays (RT-qPCR) for detecting SARS-CoV-2 infection in samples from individuals suspected of COVID-19 in São Paulo, Brazil. J Clin Virol Plus. 2021 Jun;1(1):100012. doi: https://doi.org/10.1016/j.jcvp.2021.100012.
Campos KR, Sacchi CT, Gonçalves CR, Pagnoca ÉVRG, Dias ADS, Fukasawa LO, Caterino-de-Araujo A. COVID-19 laboratory diagnosis: comparative analysis of different RNA extraction methods for SARS-CoV-2 detection by two amplification protocols. Rev Inst Med Trop Sao Paulo. 2021 Jun 25;63:e52. doi: https://doi.org/10.1590/S1678-9946202163052.
Yamada AY, Santos AMD, Souza AR, Campos KR, Sacchi CT, Tiba-Casas MR, Camargo CH. A clinical Pseudomonas chlororaphis isolate harboring a new blaVIM-2 borne integron, In2088, isolated from bloodstream infection in a newborn patient. Infect Genet Evol. 2021 Oct 5;96:105104. doi: https://doi.org/10.1016/j.meegid.2021.105104.
Resende PC, Naveca FG, Lins RD, Dezordi FZ, Ferraz MVF, Moreira EG, Coêlho DF, Motta FC, Paixão ACD, Appolinario L, Lopes RS, Mendonça ACDF, da Rocha ASB, Nascimento V, Souza V, Silva G, Nascimento F, Neto LGL, da Silva FV, Riediger I, Debur MDC, Leite AB, Mattos T, da Costa CF, Pereira FM, Dos Santos CA, Rovaris DB, Fernandes SB, Abbud A, Sacchi C, Khouri R, Bernardes AFL, Delatorre E, Gräf T, Siqueira MM, Bello G, Wallau GL. The ongoing evolution of variants of concern and interest of SARS-CoV-2 in Brazil revealed by convergent indels in the amino (N)-terminal domain of the spike protein. Virus Evol. 2021 Aug 14;7(2):veab069. doi: https://doi.org/10.1093/ve/veab069.
Dos Santos CA, Hernandes RT, Cunha MPV, Nagamori FO, Gonçalves CR, Sacchi CT, Tiba-Casas MR, Camargo CH. Two Novel Mutations Associated with Polymyxin-B Resistance in a Pandemic Lineage of Uropathogenic Escherichia coli of the Sequence Type 69. Chemotherapy. 2021;66(3):92-98. doi: https://doi.org/10.1159/000517817.
Dos Santos CA, Cunha MPV, Bertani AMJ, de Almeida EA, Gonçalves CR, Sacchi CT, de Paiva JB, Camargo CH, Tiba-Casas MR. Detection of multidrug- and colistin-resistant Salmonella Choleraesuis causing bloodstream infection. J Antimicrob Chemother. 2020 Jul 1;75(7):2009-2010. doi: https://doi.org/10.1093/jac/dkaa076.
Mustapha MM, Lemos APS, Griffith MP, Evans DR, Marx R, Coltro ESF, Siebra CA, Timm L, Ribeiro H, Monteiro A, Pasculle AW, Marsh JW, Van Tyne D, Harrison LH, Sacchi CT. Two Cases of Newly Characterized Neisseria Species, Brazil. Emerg Infect Dis. 2020 Feb;26(2):366-369. doi: https://doi.org/10.3201/eid2602.190191.
Guerra JM, Fernandes NCCA, Morales Dos Santos AL, Barrel JSP, Petri BSS, Milanelo L, Tiba-Casas MR, Liserre AM, Gonçalves CR, Sacchi CT, Catão-Dias JL, Camargo CH. Hypervirulent Klebsiella pneumoniae as Unexpected Cause of Fatal Outbreak in Captive Marmosets, Brazil. Emerg Infect Dis. 2020 Dec;26(12):3039-3043. doi: https://doi.org/10.3201/eid2612.191562.
Tavares LCB, Cunha MPV, de Vasconcellos FM, Bertani AMJ, de Barcellos TAF, Bueno MS, Santos CA, Sant'Ana DA, Ferreira AM, Mondelli AL, Montelli AC, Sadatsune T, Sacchi CT, Gonçalves CR, Tiba-Casas MR, Camargo CH. Genomic and Clinical Characterization of IMP-1-Producing Multidrug-Resistant Acinetobacter bereziniae Isolates from Bloodstream Infections in a Brazilian Tertiary Hospital. Microb Drug Resist. 2020 Nov;26(11):1399-1404. doi: https://doi.org/10.1089/mdr.2019.0210.
de Lemos APS, Sacchi CT, Gonçalves CR, Camargo CH, Andrade AL. Genomic surveillance of Neisseria meningitidis serogroup B invasive strains: Diversity of vaccine antigen types, Brazil, 2016-2018. PLoS One. 2020 Dec 21;15(12):e0243375. doi: https://doi.org/10.1371/journal.pone.0243375.
Jesus JG, Sacchi C, Candido DDS, Claro IM, Sales FCS, Manuli ER, Silva DBBD, Paiva TM, Pinho MAB, Santos KCO, Hill SC, Aguiar RS, Romero F, Santos FCPD, Gonçalves CR, Timenetsky MDC, Quick J, Croda JHR, Oliveira W, Rambaut A, Pybus OG, Loman NJ, Sabino EC, Faria NR. Importation and early local transmission of COVID-19 in Brazil, 2020. Rev Inst Med Trop Sao Paulo. 2020;62:e30. doi: https://doi.org/10.1590/s1678-9946202062030.
Tiba-Casas MR, Sacchi CT, Gonçalves CR, Almeida EA, Soares FB, de Jesus Bertani AM, Fernandes SA, de Paula Eduardo MB, Camargo CH. Molecular analysis of clonally related Salmonella Typhi recovered from epidemiologically unrelated cases of typhoid fever, Brazil. Int J Infect Dis. 2019 Apr;81:191-195. doi: https://doi.org/10.1016/j.ijid.2019.02.009.
Zanella RC, Brandileone MCC, Almeida SCG, de Lemos APS, Sacchi CT, Gonçalves CR, Gonçalves MG, Fukasawa LO, Saraiva MD, Rangel LF, Cunha JLL, Rotta TCA, Douradinho C, Jacob-Filho W, Minamisava R, Andrade AL. Nasopharyngeal carriage of Streptococcus pneumoniae, Haemophilus influenzae, and Staphylococcus aureus in a Brazilian elderly cohort. PLoS One. 2019 Aug 22;14(8):e0221525. doi: https://doi.org/10.1371/journal.pone.0221525.
Ouattara M, Whaley MJ, Jenkins LT, Schwartz SB, Traoré RO, Diarra S, Collard JM, Sacchi CT, Wang X. Triplex real-time PCR assay for the detection of Streptococcus pneumoniae, Neisseria meningitidis and Haemophilus influenzae directly from clinical specimens without extraction of DNA. Diagn Microbiol Infect Dis. 2019 Mar;93(3):188-190. doi: https://doi.org/10.1016/j.diagmicrobio.2018.10.008.
Whaley MJ, Jenkins LT, Hu F, Chen A, Diarra S, Ouédraogo-Traoré R, Sacchi CT, Wang X. Triplex Real-Time PCR without DNA Extraction for the Monitoring of Meningococcal Disease. Diagnostics (Basel). 2018 Aug 30;8(3):58. doi: https://doi.org/10.3390/diagnostics8030058.
Relatório Executivo 2017-2022
-
23687 KB
LEIAL 2017-2022.pdf
Formato: PDF
visualizar download -
1435 KB
Vigilância Genômica de S. Typhi, V. cholerae e N. meningitidis
Formato: PDF
visualizar download -
1658 KB
Sequenciamento do vírus Chikungunya
Formato: PDF
visualizar download -
3116 KB
Dengue 1 - Monitoramento Genômico
Formato: PDF
visualizar download -
2947 KB
Vigilância da Difteria.pdf
Formato: PDF
visualizar download -
2199 KB
Dengue 2 - Monitoramento Genômico
Formato: PDF
visualizar download -
4601 KB
Primeiro Caso de Monkeypox
Formato: PDF
visualizar download -
2354 KB
Primeira Epizootia por Febre Amarela
Formato: PDF
visualizar download -
1826 KB
Febre Amarela - Caso em Humano
Formato: PDF
visualizar download -
570 KB
Investigação Genômica: E. coli hipervirolentas
Formato: PDF
visualizar download -
1863 KB
Detecção e Sequenciamento de Dengue 3 da Vigilância Sentinela.pdf
Formato: PDF
visualizar download -
1340 KB
Sequenciamento por leituras longas na plataforma PacBio
Formato: PDF
visualizar download -
2923 KB
Primeiros Diagnósticos
Formato: PDF
visualizar download -
2872 KB
Primeiro Sequenciamento
Formato: PDF
visualizar download -
6840 KB
Reinfecção pelo SARS-CoV-2
Formato: PDF
visualizar download -
1267 KB
Detecção da linhagem P.1 (variante gamma)
Formato: PDF
visualizar download -
1420 KB
Operação Acolhida
Formato: PDF
visualizar download -
5182 KB
Detecção da linhagem B.1.1617.2 (variante delta)
Formato: PDF
visualizar download -
2944 KB
Monitoramento Copa América
Formato: PDF
visualizar download -
1341 KB
MANUAL PROCEDIMENTOS OBRIGATÓRIOS PARA O SEQUENCIAMENTO DE ARBOVIROSES NA RNSG - MINISTÉRIO DA SAÚDE
Formato: PDF
visualizar download

